太阳成集团tyc122cc/学院新闻 2021-07-19 19:50:17 来源:太阳成集团tyc122cc 点击: 收藏本文
近日,我院罗琼教授团队与生物光子学研究院青年英才孟露明博士联合在《Nature Communications》(影响因子:12.121)在线发表题为“Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data”的学术论文。孟露明博士为论文第一作者兼通讯作者,我院2020届硕士研究生王晨曦为第二作者。
2020年国家基金委项目指南中优先支持方向之一是,能为生命科学研究提供全新的思路和理念,推进实现生命过程(或功能)研究的可视、可控、可创造的化学研究。本工作是化学与生物的交叉研究,它结合计算化学方法对哺乳动物染色质的三维结构实现高精度的可视化。
哺乳动物的染色质线性长度可达2米,它折叠在直径为10-20微米的细胞核中形成复杂的三维结构,该结构对基因表达、细胞分化以及疾病发生等生命过程具有重要的调控作用。如何得到单细胞的染色质三维结构是生命科学中的一个核心问题。
研究单细胞全基因组染色质三维结构的主流实验手段之一是单细胞Hi-C技术。它能够测量得到线性距离(或者说序列距离)远但空间距离近的染色质片段对。利用单细胞Hi-C技术研究染色质三维结构的难点在于,(1)Hi-C实验给出的染色质三维结构信息是二维的,即间接的;(2)Hi-C技术不能将每一对在空间上相互靠近的染色质片段都标识出来,意味着Hi-C数据给出的结构信息是不完整的;(3)Hi-C数据是有噪声的。如何从单细胞 Hi-C 数据中准确地构建出高分辨率的染色质三维结构是当代生物学的主要挑战之一。
本研究工作结合统计学的贝叶斯理论和化学的分子模拟方法建立了Si-C方法,该方法成功地从哺乳动物的单细胞 Hi-C数据中构建出全基因组三维结构。除了Si-C方法,文献中报道的具有相似功能的方法只有一个,即牛津大学Laue课题组于2017年在Nature上报道的 NucDynamics方法(Nature 544, 59-64 (2017)) 。与NucDynamics方法相比,Si-C方法的优势有三点:(1) 建模速度快,是NucDynamics方法的两倍;(2)在相同的实验数据下,Si-C能够得到更高精度的结构,比NucDynamics方法高出近两个数量级;(3)Si-C得到的结构具有明确的统计学意义。
如果将单细胞Hi-C实验技术提供的结构信息比喻为二维码,本研究建立的Si-C方法就是一个高效且准确的扫码器。Si-C方法提供的高精度染色质三维结构将大大降低三维结构与生物功能相关性研究的门槛。Si-C方法在癌症的治疗与预防、细胞再生以及精准医疗等领域具有广泛的应用前景。
论文链接:https://www.nature.com/articles/s41467-021-24662-z

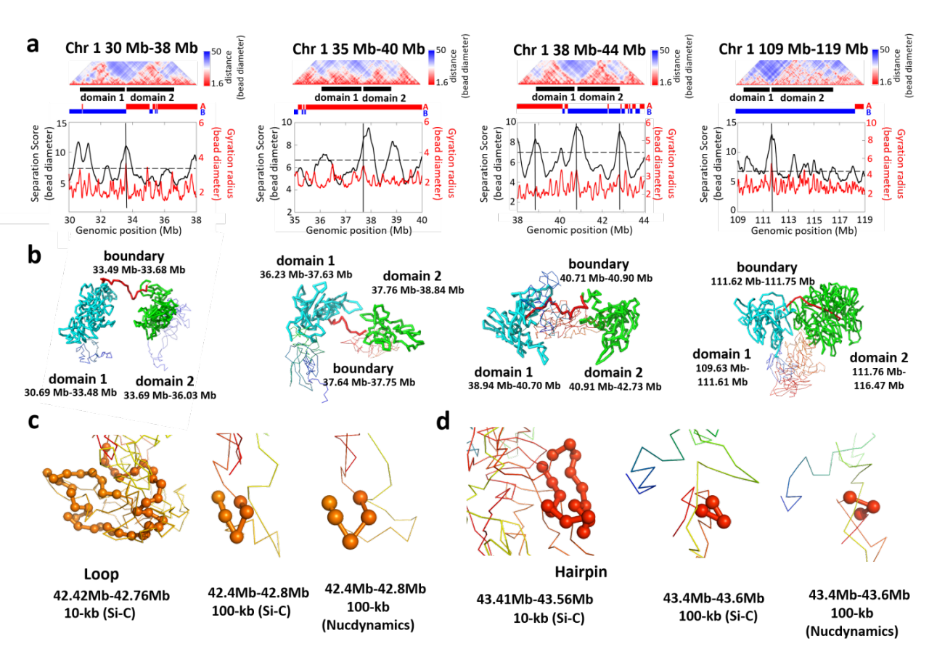
Fig. 5: Small-scale 3D structure at 10-kb resolution.
标签: